こんにちは。
“エコモット アドベントカレンダー10日目” 担当 岩津です。
最近はグラフ生成にはまってます。(色んな意味で)
今回はMatplotlibを使った手軽なグラフ作成環境としてSingularityを使うことを考えてみます。
Singularityって何?
こちらを参照ください。(今回の記事はGPU関係ないです)
『GPUユーザーは、すぐにdockerからsingularityに切り替えるべきだと思った8つのこと』
なお、Singularityの導入は完了している前提です。
(今回はWindows10上のHyper-VにUbuntu19.10を構築してます)
Singularityはコンテナ仮想化プラットフォームですが、同じくコンテナ仮想化であるDockerのイメージをコンバートして利用できます。
使い方はDockerリポジトリからpullするだけです。
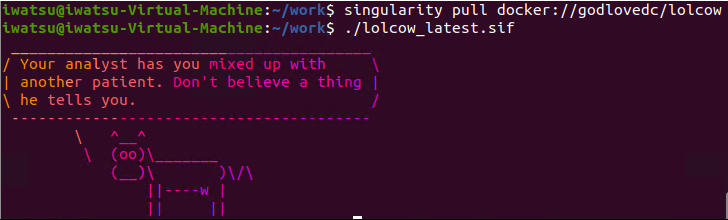
Singularityはイメージファイルを直接実行できるので、上記の図のようにまるで実行ファイルのように扱えます。
今回はグラフ作成環境のベースとしてDocker Hubからjupyter notebookのイメージを利用します。
イメージがダウンロードできたら
|
1 2 |
$ ./datascience-notebook_latest.sif |
で実行してみます。
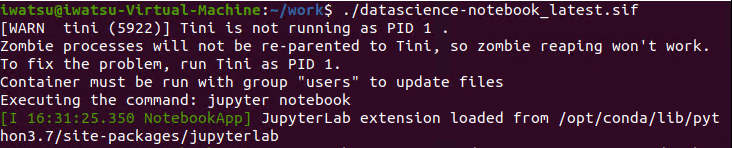
(…中略…)
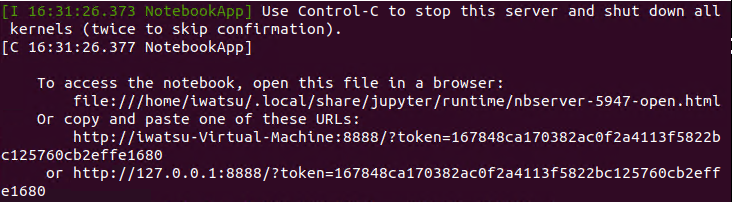
このイメージの場合、jupyter notebookのサーバーが普通に起動しました。
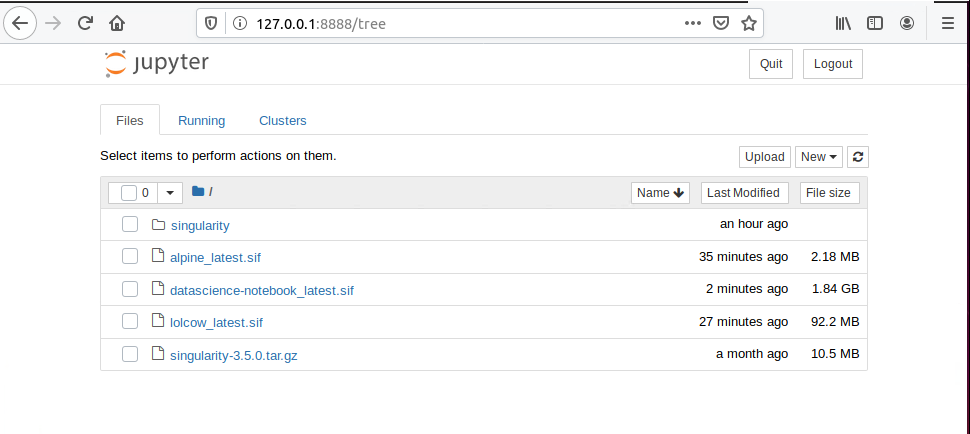
Singularityでは実行時ディレクトリを自動でマウントしているので、すでにディレクトリにあるファイルが見えています。
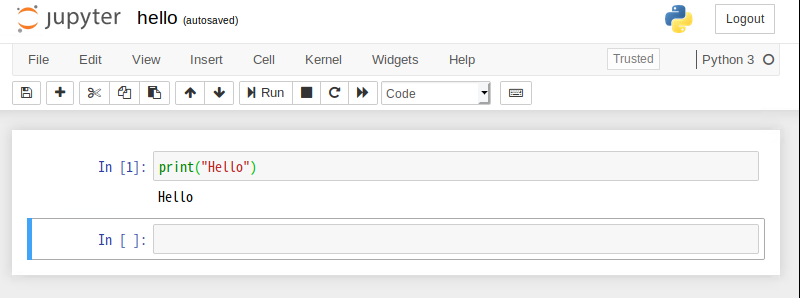
まずは試しに簡単なプログラムを書きました。
(実行できてます)
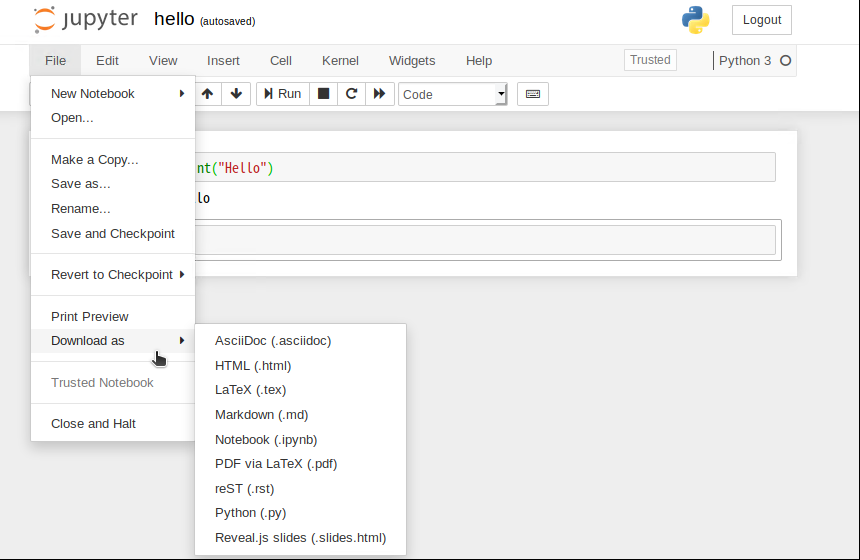
これをコマンドで実行できるように.pyで、イメージを実行したディレクトリに保存します。ファイル名はhello.pyです。
見えてますね。

|
1 2 |
$ singularity exec ./datasience-notebook_latest.sif python ./hello.py |
Singularityで実行する場合、通常execコマンドでイメージパスと実行コマンドを指定すれば良いです。
ここまでできたので次はnotebookでMatplotlibを使ってグラフを書いてみます。(Matplotlibのサンプルを利用)
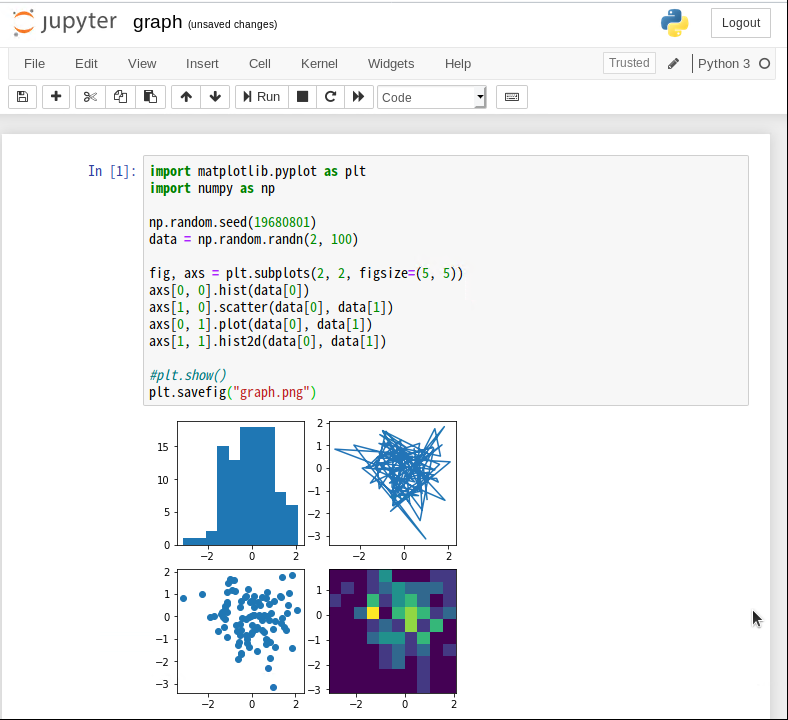
最後にsavefigでファイルを保存するように変更しています。
notebookで生成した画像ファイルを削除しておいて、先程と同じように.pyに変換したファイルを作ります。(ファイル名はgraph.py)
|
1 2 |
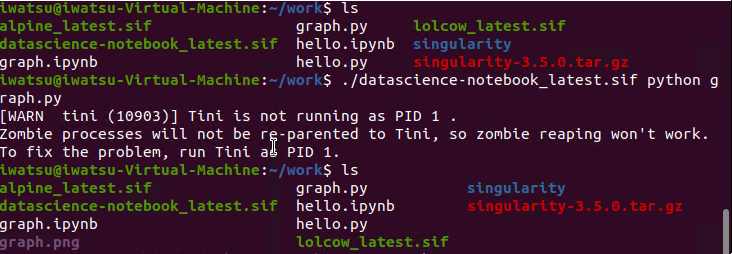
$ ./datasience-notebook_latest.sif python ./graph.py |
今回のイメージの場合、イメージを直接実行するときの引数で実行したいコマンドを渡すことができます。(正確にはrunscriptの設定によりますが割愛)
グラフ画像のgraph.pngが生成されていることが確認できました。
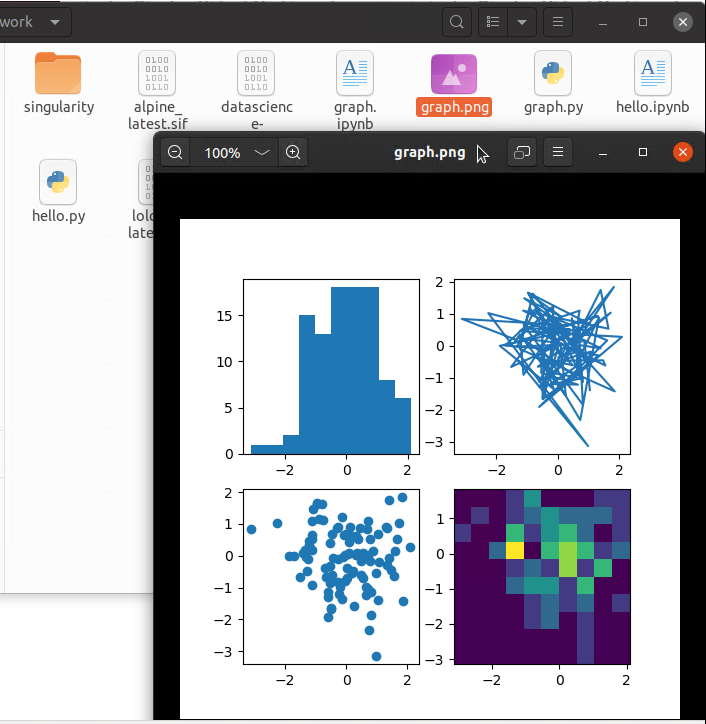
ビューワーでも確認。
Singularityがローカルのディレクトリを自動マウントしてくれるおかげで、手軽に.pyファイルをコンテナに渡す事ができます。
jupyter notebookと同じイメージを使ってグラフ生成プログラムをバッチ実行することができるので、グラフを作りやすい環境ができました。





